[사이언스] 염기교정 유전자가위의 안전성 높일 실마리 찾았다인공지능 이용한 점돌연변이 질환 관련 염기교정 유전자가위의 효율 및 교정결과 예측
국내연구진이 딥러닝 기법을 이용해 점돌연변이 교정을 위한 유전자가위의 안전성을 높일 수 있는 실마리를 찾아냈다.
한국연구재단은 연세대학교 약리학교실 김형범 교수 연구팀이 염기교정 유전자가위의 염기교정 효율과 교정결과를 예측할 수 있는 인공지능 프로그램을 개발했다고 9일 밝혔다.
대를 거듭해 보존되는 ‘원본’인 유전자에 대한 외부개입인 만큼 유전자가위는 편집효율이 일정 수준 이상이어야 하고, 원치 않는 다른 염기의 편집이 일어나지 않아야 한다. 즉 효율과 정확성, 두 가지 모두를 충족해야 한다.
특정 염기를 바꿔(C->T 혹은 A->G) 주는 염기교정 유전자가위는 유전질환을 일으킬 수 있는 점돌연변이를 바로잡거나 반대로 유전질환을 가진 동물모델을 얻기 위한 매력적인 도구가 될 수 있다.
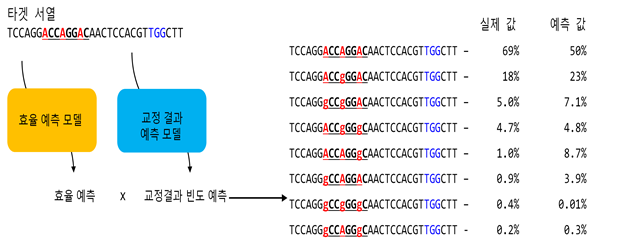
하지만 고정된 한 자리에서 편집이 일어나는 것이 아니라 일정 범위 안에 같은 종류의 염기가 여럿이 존재한다면, 원하지 않는 염기가 편집될 수 있다. 때문에 위치별 편집빈도를 예측, 가장 안전한 유전자가위를 선별하는 과정이 필수적이었다.
이에 연구팀은 다양한 염기교정 유전자가위를 만들고, 각각의 효율과 결과물의 빈도에 대한 빅데이터를 확보, 딥러닝으로 분석하여 염기교정 결과예측 프로그램(DeepBaseEditor)을 개발하였다.
나아가 이 프로그램을 이용해 23,479개의 점돌연변이 유전질환 가운데 염기교정 범위 내 표적염기가 1개이면서 효율(5%이상)이 높을 것으로 예상되어 염기교정 유전자가위로 유전자편집을 시도해볼 수 있는 질환으로, 낭포성 섬유증(cystic fibrosis) 등 3,058개 가량의 점돌연변이 유전질환을 1차적으로 선별해냈다.
점돌연변이 유전질환 가운데 상당수(약 19,505개)가 염기교정이 일어날 수 있는 범위에 동일 염기(아데닌 또는 시토신)가 2개 이상 자리하고 있어, 원하지 않는 위치에서의 편집확률을 미리 예측하는 것이 중요했다.
연구팀은 이들 19,505개의 유전질환 중 약 4,274개의 유전자에 대해 효율(5%이상)이 높으면서, 다른 염기의 변이가 잘 일어나지 않을 것으로 예측하였다. 선별된 유전질환들은 표적염기가 2개 이상으로 추가적인 변이가 발생할 수 있어 사용이 어려웠던 유전질환들이었다. 본 연구를 통해 위 질환들은 추가적인 변이 가능성이 낮을 것으로 예상되어 염기교정 유전자가위를 사용이 가능할 것으로 기대된다.
연구팀은 향후 본 예측 프로그램을 이용해 선별된 유전자가위를 활용, 질환 동물모델 수립연구를 추진할 계획이다.
이번 연구성과는 생명공학 분야 국제학술지 네이처바이오 테크놀로지(Nature Biotechnology)에 7월 7일(한국시간)에 게재되었다.
한편, 6월말 김형범 연세대 교수가 Nature Biotechnology에 발표한 논문이 보도된 바 있다. 기존 성과는 일반 유전자가위(크리스퍼 유전자가위의 다양한 개량버전들을 예측, 비교하는 프로그램)에 대한 것이며, 이번 성과는 염기교정 유전자가위(염기교정 유전자가위 적용시 효율이 높고 다른 변이가능성 낮은 점돌연변이 예측 프로그램)에 대한 것이다.
이 기사 좋아요
<저작권자 ⓒ 특허뉴스 무단전재 및 재배포 금지>

댓글
염기교정,유전자가위,인공지능,점돌연변이,딥러닝,김형범교수,유전질환,유전자편집 관련기사목록
|
많이 본 기사
|